
下面我们逐一介绍单细胞测序技术:单细胞测序技术概念及发展历程、单细胞测序技术操作流程、单细胞全基因组测序技术、单细胞全转录组测序技术、单细胞测序技术的应用。
单细胞测序技术
单细胞测序(Single cell sequencing,SCS)技术是指在单个细胞水平上对转录组或基因组进行扩增并测序,以检测单细胞在基因组学、转录组学、表观组学和蛋白组学等多个组学的数据。主要涉及:单细胞基因组测序、单细胞转录组测序和单细胞表观基因组测序。
单细胞基因组测序(图1A):是将分离的单个细胞的微量全基因组DNA进行扩增,获得高覆盖率的完整的基因组后进行高通量测序,用于揭示单细胞中的遗传变异,如单核苷酸变异(SNVs)、拷贝数变异(CNVs)和基因组结构变异(SVs),细胞群体差异和细胞进化关系。
单细胞转录组测序(图1B):是将分离的单个细胞的微量全转录组RNA进行扩增后进行高通量测序,用于在单细胞中生成基因表达、基因融合和选择性剪接的图谱,此技术被认为是截至 2020 年定义细胞状态和表型的金标准。[1]
单细胞表观基因组测序(图1C):是检测DNA序列不变的情况下表型的可遗传变化,包括DNA甲基化、组蛋白修饰、染色质可及性等。在真核生物中,5-甲基胞嘧啶(5mC) 在基因组中广泛分布,并通过抑制转座因子在调节基因表达中发挥重要作用[2]。通过对单个细胞中的 5mC 进行测序,可以揭示来自单个组织或群体的遗传相同细胞的表观遗传变化如何产生具有不同表型的细胞。单细胞亚硫酸氢盐测序是DNA甲基化研究的金标准。
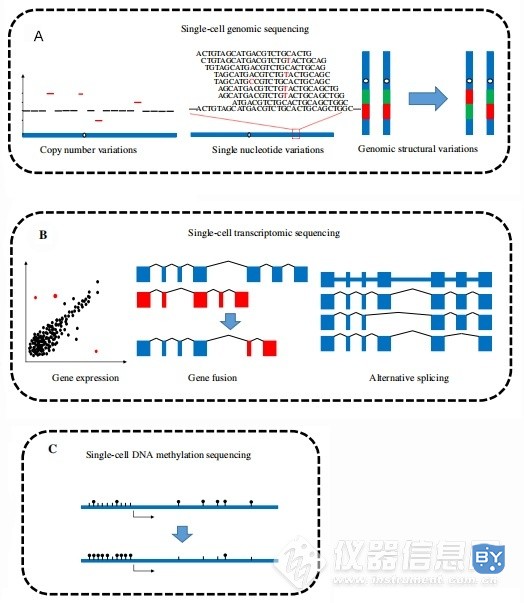
图1 单细胞测序技术应用范围示意图[3]
A:单细胞基因组测序应用范围;B:单细胞转录组测序应用范围;C:单细胞DNA甲基化测序应用范围;
为什么要做单细胞测序呢?
多细胞生物在细胞的分裂和分化过程中必然会带来不同细胞间的差异,形成遗传信息的异质性。传统的检测方法获得的信息来自于数百万甚至更多细胞的混合样本,因此得到的结果反映的是一群细胞中信号的平均值,或者只代表其中占优势数量的细胞信息,导致不同细胞间异质性信息被忽视。而单细胞测序可以检测单个细胞异质性、识别稀有细胞、揭示细胞间差异情况。[4]
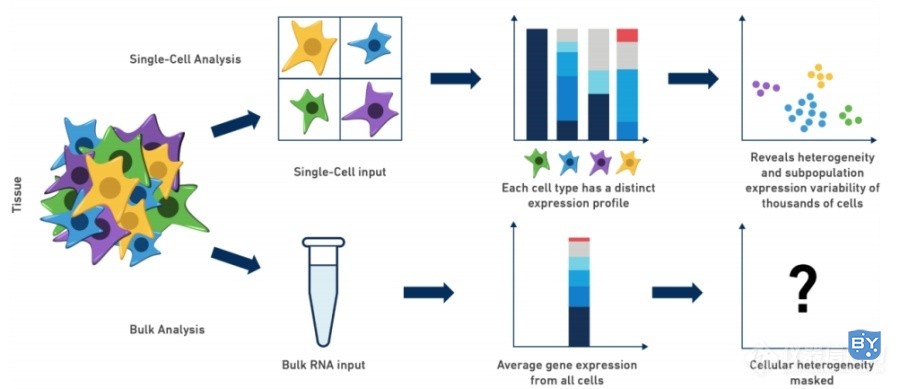
图2 单细胞测序(上)与传统混合细胞测序(下)对比示意图
单细胞测序技术发展
2009年汤富酬等完成首例哺乳动物单细胞RNA转录组测序后,单细胞测序经历了十几年突飞猛进的发展,同时,随着测序技术的更新迭代,各厂商基于不同检测原理开发出的单细胞分析系统不断推陈出新,单细胞测序逐渐实现了从低通量到高通量检测的转变。2017年“人类细胞图谱计划(Human Cell Atlas,HCA)”的正式公布,是高通量单细胞研究产业化的重要里程碑。
![图3 单细胞研究发展重大历程[5] 图3 单细胞研究发展重大历程[5]](/static/upload/image/20220726/1658799116593126.jpg)
图3 单细胞研究发展重大历程[5]
单细胞测序技术流程
最初单细胞测序是采用不同方法将单个细胞分离出来,独立构建成文库进行测序。但此法分离细胞通量低(仅检测数十个细胞且不足以反应真实情况)且成本较高。
随着测序技术的发展,出现了基于标签(barcode)的单细胞识别技术,即不需要分离单个细胞,仅需对每个细胞加上单独的标签序列,通过一次建库测序即可,此方法使得单细胞测序进入了高通量时代,单细胞分离和测序的成本大大降低。
与传统混合细胞测序不同的是,单细胞测序起始样本中核酸含量极低,需要对筛选出的细胞扩增后才能满足后期测序实验,目标是在尽量减少序列扩增偏差的前提下增加核酸总量利于后续分析。单细胞测序技术操作流程包括:样本细胞筛选、核酸提取及扩增、测序文库构建、测序和数据分析。
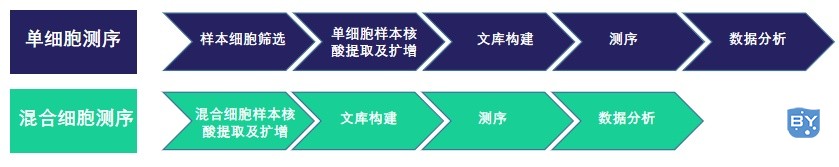
图4 单细胞测序(上)与传统混合细胞测序(下)技术流程对比示意图
参考文献
[1] Tammela,Tuomas; Sage,Julien (2020). "Investigating Tumor Heterogeneity in Mouse Models". Annual Review of Cancer Biology. 4(1):99–119.doi:10.1146/annurev-cancerbio-030419-033413.
[2] Zemach A, McDaniel IE, Silva P, Zilberman D (May 2010). "Genome-wide evolutionary analysis of eukaryotic DNA methylation". Science. 328 (5980): 916-9. Bibcode:2010Sci...328..916Z. doi:10.1126/science.1186366.
[3] Jialong Liang , Wanshi Cai , Zhongsheng Sun.Single-Cell Sequencing Technologies: Current and Future[J].Journal of Genetics and Genomics 41 (2014) 513-528
[4] Eberwine J, Sul JY, Bartfai T, Kim J ,The promise of single-cell sequencing[J]. Nature Methods. 2014,11 (1): 25–7. doi:10.1038/nmeth.2769
[5] 基因慧《2020单细胞行研报告》
 文章推荐
文章推荐